Clustering ARN - Programmation Parallèle
Chaîne Floyd-PAM pour le clustering ARN, parallélisée en MPI et OpenMP
C++MPIOpenMPProgrammation DynamiqueAlgorithmes de Graphes
Voir le codeProjet académique de Master réalisé dans le cadre du cours de Programmation Parallèle à l'Université d'Orléans. L'objectif était d'implémenter Floyd-Warshall et PAM (k-médoïdes) avec une approche hybride MPI+OpenMP, puis d'appliquer cette chaîne au clustering de séquences d'ARN.
Le pipeline part d'un graphe pondéré construit à partir de distances entre séquences (Hamming pour la rapidité, ou alignement Needleman-Wunsch pour plus de précision), calcule les plus courts chemins, puis forme k partitions. L'implémentation utilise MPI pour la distribution en mémoire et OpenMP pour la parallélisation des calculs locaux, s'appuyant sur un découpage en blocs et des communicateurs ligne/colonne pour limiter les échanges. Cette approche hybride améliore significativement la scalabilité. Les tests montrent des gains significatifs sur grands graphes et une forte sensibilité aux paramètres n, k et ε.
Construction du graphe de similarité
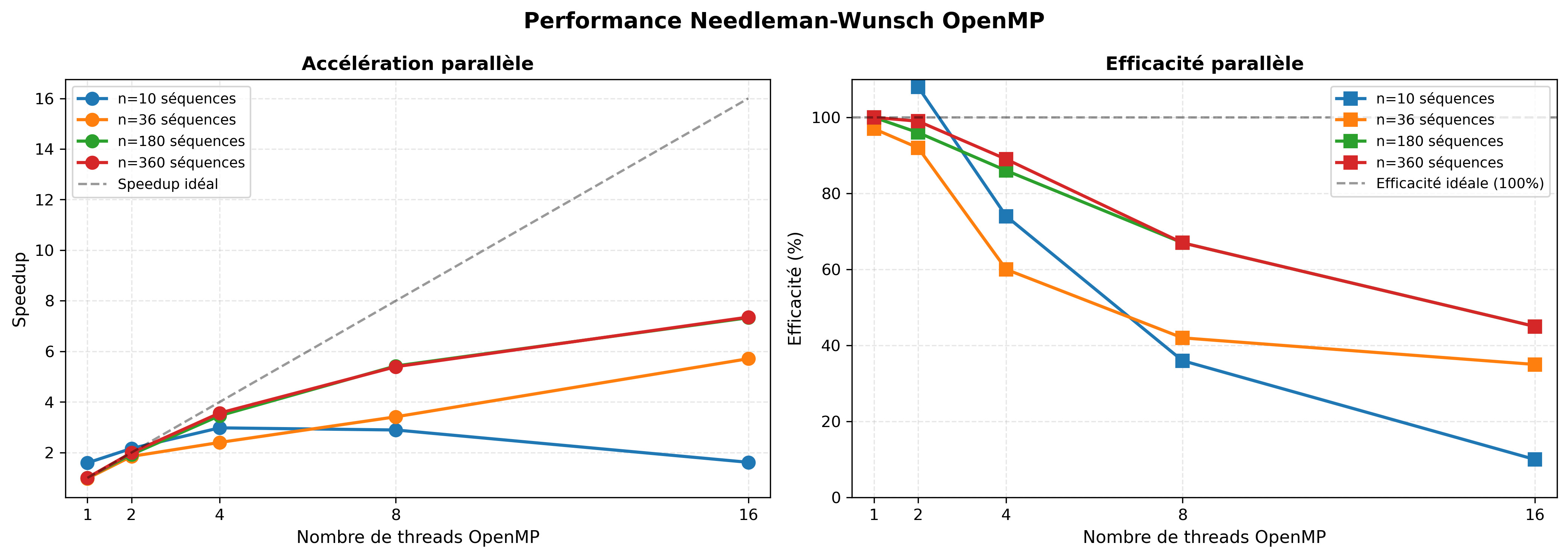
La première étape construit la matrice d'adjacence des séquences ARN. Pour des jeux de données rapides, la distance de Hamming est utilisée ; pour des comparaisons plus fines, un alignement Needleman-Wunsch avec pénalités de gaps est appliqué.
Le calcul est parallélisé en répartissant les lignes de la matrice entre processus (complexité O(n²·L/p)). Sur certaines configurations, une variante OpenMP accélère l'alignement local tout en conservant la précision.
Floyd-Warshall MPI (décomposition par blocs)
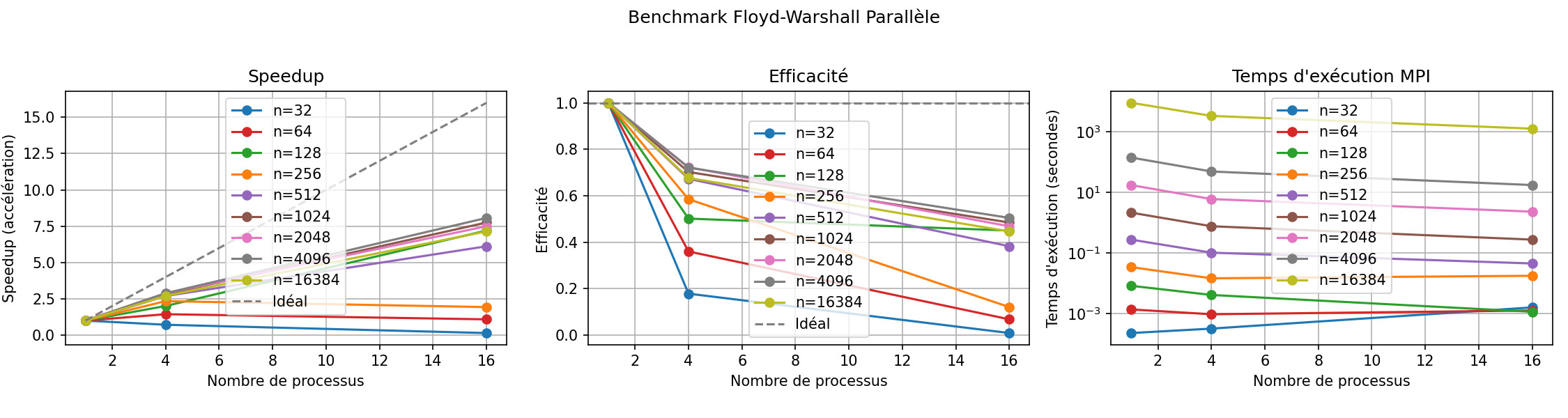
Cette étape calcule la matrice des plus courts chemins sur un graphe pondéré. La matrice D est découpée en blocs b×b et distribuée sur une grille 2D de processus (q×q avec q = n/b).
À chaque itération k, le bloc diagonal est mis à jour puis diffusé ; la ligne et la colonne k sont ensuite mises à jour et redistribuées, permettant aux autres blocs de se calculer en parallèle. Des communicateurs MPI ligne/colonne (MPI_Comm_split) réduisent le coût des échanges.
PAM k-médoïdes en MPI
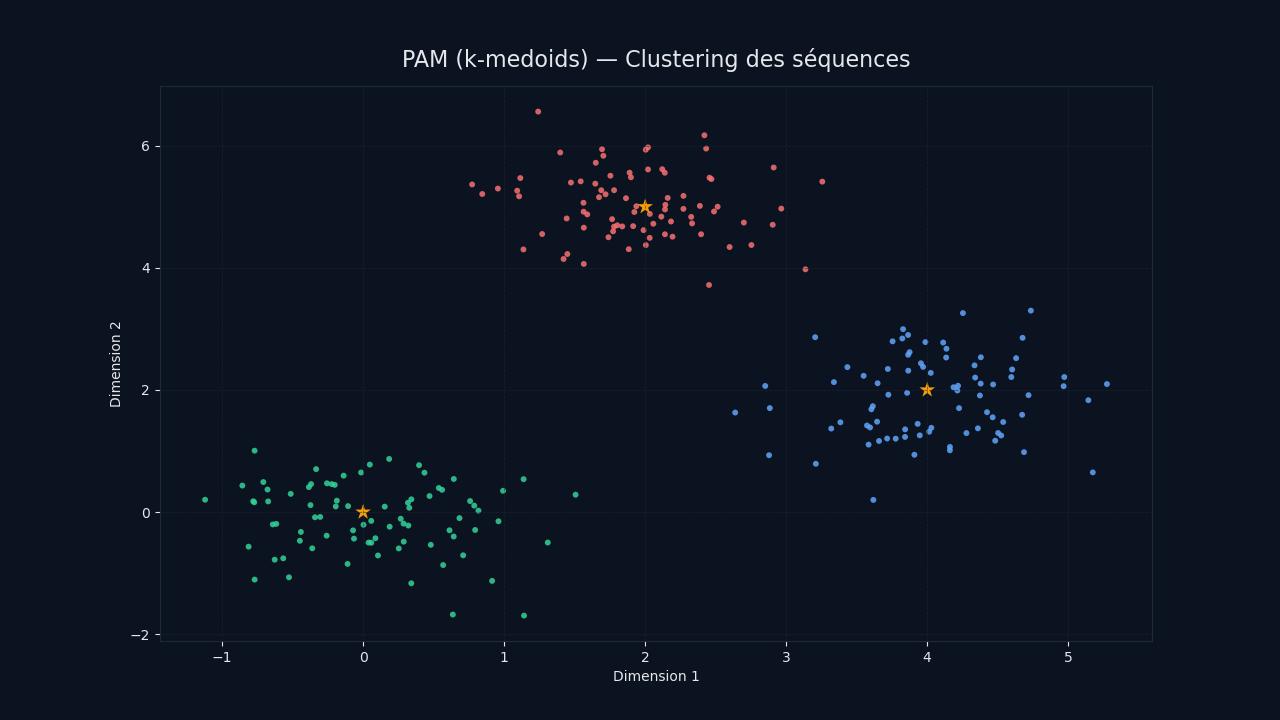
La phase BUILD initialise k médoïdes, puis la phase SWAP teste des échanges médoïde ↔ non-médoïde pour minimiser le coût total. Chaque test est indépendant : les candidats sont distribués de manière cyclique entre processus.
La matrice des distances est diffusée une seule fois (MPI_Bcast), et chaque itération utilise un MPI_Allreduce (MINLOC) pour sélectionner le meilleur swap. La complexité par itération est en O(n²·k/p), et l'initialisation aléatoire rend le résultat non déterministe.
Application ARN & sensibilité aux paramètres
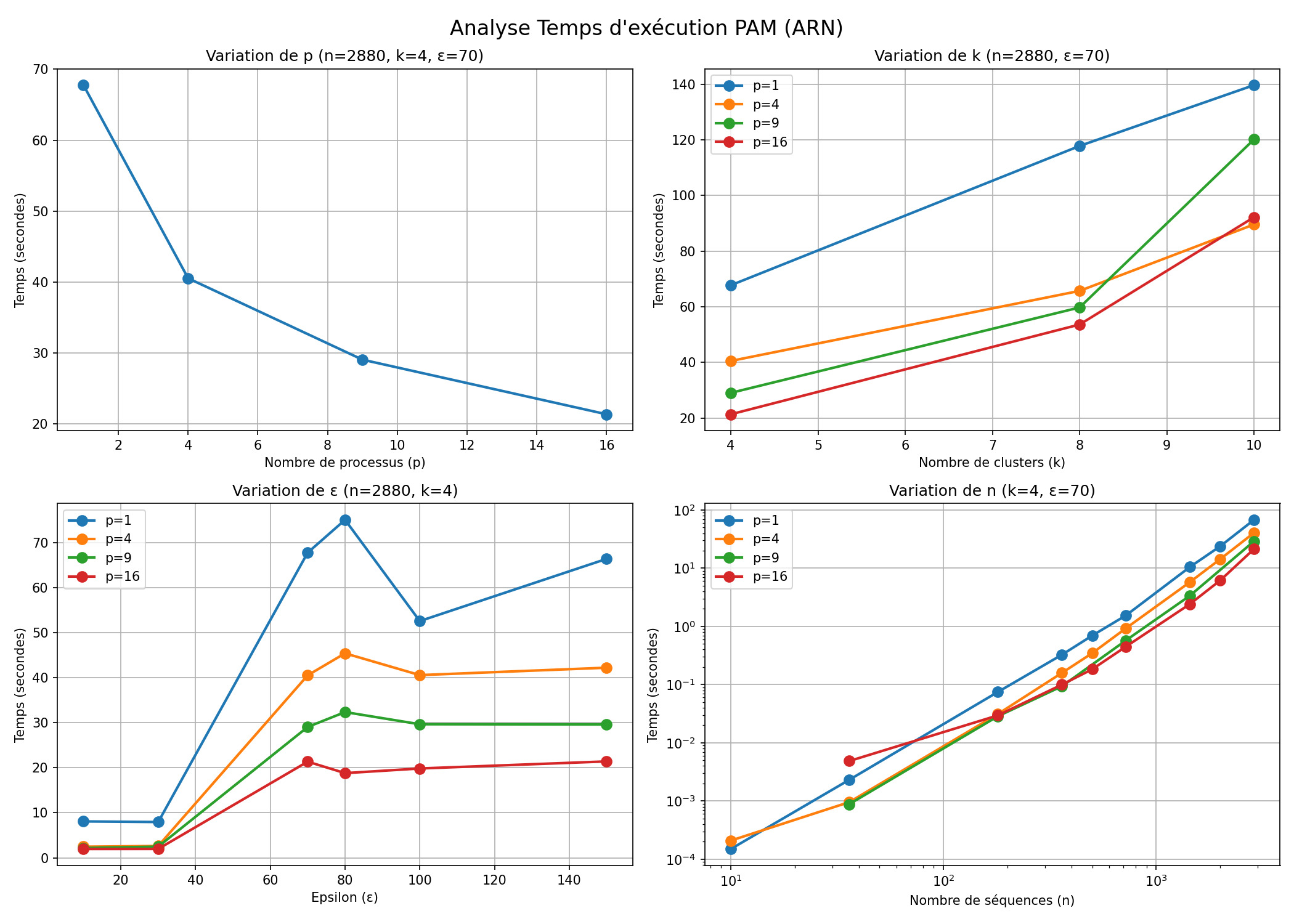
Le pipeline complet enchaîne : distances (Hamming/Needleman) → Floyd-Warshall → PAM pour obtenir k clusters. Les expérimentations montrent un impact fort de ε (seuil de création d'arêtes) : faible ε → graphe sparse et temps réduit ; ε moyen → graphe dense et temps maximal ; ε élevé → graphe quasi complet et convergence plus rapide.
Le temps croît linéairement avec k et de façon cubique avec n (Floyd). Pour de petites tailles, l'overhead MPI domine ; dès n ≥ 500, la parallélisation devient réellement bénéfique (gain notable pour n = 2000).
Résultats & scalabilité (MPI/Hybride)
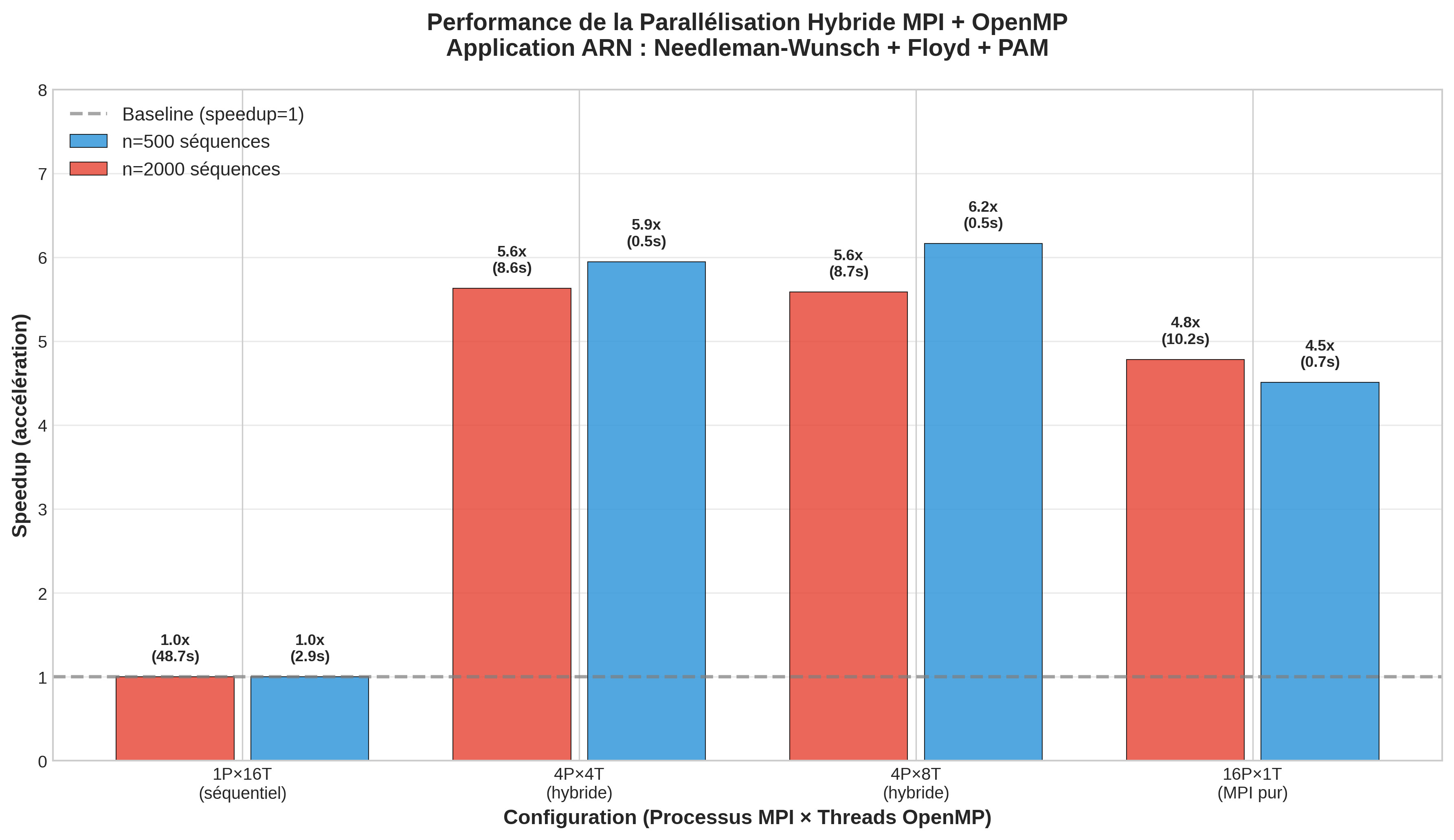
Sur Floyd, le speedup passe de 1× (p=1) à 2,69× (p=4) et 6,12× (p=16) pour n=512. Sur des tailles plus grandes (n=16384), le temps séquentiel descend de 2h29 à environ 21 minutes avec 16 processus.
Dans la variante hybride MPI+OpenMP, les tests confirment qu'une configuration équilibrée (ex. 4P×8T) offre le meilleur compromis entre calcul local et communications.